近日,生物所创新团队联合攻关开发了基于人工智能的水稻染色质开放性在线预测模型,为水稻染色质开放性及其转录调控研究提供了新思路。相关成果发表在《遗传学和基因组学杂志(Journal of Genetics and Genomics)》上。
染色质物理状态由DNA折叠和缩合决定,一般分为常染色质和异染色质两种状态,但两者并不是静态不变的,而是随着生长发育、环境响应等变化动态调整。染色质开放性(chromatin accessibility)反映了染色质转录活跃程度,结合其它表观遗传修饰如DNA甲基化等信息,可以提供全基因组的基因表达调控信息。因此,识别染色质开放性区域对理解基因表达调控如何协调生长发育与对环境刺激的响应至关重要。由于受实验技术所限,大量染色质开放性区域至今仍未得到完全鉴定,因此开发基于人工智能的预测方法显得尤为重要。
本研究通过分别使用正常和热处理条件下籼粳稻的染色质开放性数据,进行智能预测模型(Smart Model for Open Chromatin region prediction, SMOC)构建。在籼粳稻模型之间的交叉验证中,预测数据和实验数据的皮尔森相关系数均在0.97以上。与其它机器学习方法进行性能比较发现SMOC具有更优性能。此外,染色质开放性和DNA甲基化6mA的关联分析结果表明,染色质开放性可能与6mA协同调控基因表达。本研究开发建立了一个染色质开放性智能预测模型(http://www.elabcaas.cn/smoc/index.html),可以快捷地进行染色质开放性预测。为染色质开放性区域鉴定和信息挖掘提供新的研究思路,结合之前建立的水稻智能数据库eRice(2020)、作物表观遗传智能预测模型SMEP(2021)等,初步形成链条式智能算法开发和模型构建,将为今后作物智能设计育种提供新的数据资源和工具。
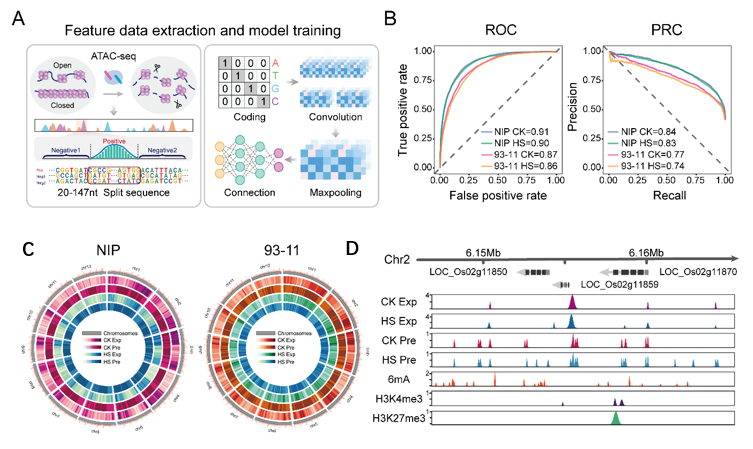
水稻染色质开放性智能预测模型
A:基于卷曲神经网络(CNN)的模型构建流程;B:模型性能评估;C和D:预测和实验数据在全基因组和局部区域的比较。
生物所博士生郭位军、硕士生刘汗青、王一凡及已毕业博士生张平贤为该论文共同第一作者,普莉研究员、田健研究员和谷晓峰研究员为共同通讯作者。相关工作得到国家自然科学基金、中央级公益性科研机构基本科研业务费等项目资助。
原文链接:
https://www.sciencedirect.com/science/article/pii/S1673852722000522?via%3Dihub


